PCA가 무엇인지 알았으니, 이번에는 iris 데이터를 가지고 실습해보도록 하겠습니다.
iris 데이터를 전처리할 pandas와 sklearn에서 iris 데이터를 가져오겠습니다.
import pandas as pd
from sklearn.datasets import load_iris
iris = load_iris()
iris_pd = pd.DataFrame(iris.data, columns=iris.feature_names)
iris_pd['species'] = iris.target
iris_pd.head()
데이터를 다시 기억해서 알아보기위해 seaborn으로 확인해보겠습니다.
import seaborn as sns
sns.pairplot(iris_pd, hue='species', height=3,
x_vars=['sepal length (cm)', 'petal width (cm)'],
y_vars=['petal length (cm)', 'petal width (cm)']
);
이전에는 못보던 옵션이 추가되었죠? x_vars, y_vars인데요, 이게 없으면 어땠었는지 기억나나요
원래 우리가 보던 pairplot은 species에 의해 색상이 구분된 이와같은 그래프였죠. ㅎㅎㅎ x_vars, y_vars로 보고싶은 feature만 구분한겁니다.
sns.pairplot(iris_pd, hue='species');
일단 scaler를 적용해보겠습니다.
from sklearn.preprocessing import StandardScaler
ss = StandardScaler()
iris_ss = ss.fit_transform(iris.data)
iris_ss[:3] #3개 데이터만 보겠습니다.array([[-0.90068117, 1.01900435, -1.34022653, -1.3154443 ],
[-1.14301691, -0.13197948, -1.34022653, -1.3154443 ],
[-1.38535265, 0.32841405, -1.39706395, -1.3154443 ]])
다시 복습차원에서 Standard Scaler를 살펴보면,
`StandardScaler`는 사이킷런(Scikit-learn) 라이브러리에서 제공하는 데이터 전처리 도구로, 데이터의 각 특성(변수)을 평균이 0이고, 분산이 1이 되도록 표준화(정규화)하는 방법입니다. `StandardScaler`는 데이터 전처리 과정에서 각 특성을 평균 0, 분산 1로 변환하는 방법입니다. 이는 머신러닝 모델이 데이터의 특성 스케일에 민감하지 않게 하고, 최적화 알고리즘이 더 안정적이고 빠르게 수렴하도록 도와줍니다.
다른 Scaler도 기억나시나요?
StandardScaler : 각 특성의 값을 평균이 0, 분산이 1이 되도록 변환합니다. 데이터의 중심을 맞추고 분산을 동일하게 조정하여, 모델이 각 특성을 균등하게 고려할 수 있게 합니다.
MinMaxScaler : 각 특성의 값을 지정된 최소값과 최대값(기본값: 0과 1) 사이로 변환합니다. 모든 데이터가 같은 범위 내에 위치하게 되어 특성의 크기 차이가 줄어듭니다. 이상치에 민감할 수 있습니다. 이상치가 있는 경우 스케일링 결과가 왜곡될 수 있습니다.
RobustScaler : 중앙값(median)과 IQR(Interquartile Range, 1사분위수와 3사분위수의 차이)을 사용하여 데이터를 스케일링합니다. 이상치(outlier)에 덜 민감합니다. 중앙값과 IQR을 사용하므로 데이터의 분포가 비대칭인 경우에도 효과적입니다.
다시 기억해주는 것이 좋습니다.
강의에서는 pca를 받아서 fit 시키고 transform 시키는 과정을 get_pca_data()라는 함수를 만들어 적용합니다.
from sklearn.decomposition import PCA
def get_pca_data(ss_data, n_components=2):
pca = PCA(n_components=n_components)
pca.fit(ss_data) #scaling이 적용된 데이터라는 뜻입니다.
return pca.transform(ss_data), pca #pca도 필요하므로 같이 출력하겠습니다.
위에 scaling 시킨 iris_ss를 넣고, 주성분을 2개로 해서 get_pca_data()함수를 통해 fit, transform 시키겠습니다.
iris_pca, pca = get_pca_data(iris_ss, 2)
iris_pca.shape(150, 2)
pca.mean_array([-1.69031455e-15, -1.84297022e-15, -1.69864123e-15, -1.40924309e-15])
feature가 4개다 보니까 중앙값도 4개로 표시되네요.
pca.components_array([[ 0.52106591, -0.26934744, 0.5804131 , 0.56485654],
[ 0.37741762, 0.92329566, 0.02449161, 0.06694199]])
행이 2개니까, 벡터가 총 2개죠.
pca.explained_variance_array([2.93808505, 0.9201649 ])
설명력도 확인해보겠습니다. 그치만 역시 ratio로 보는게 보기 쉽네요.
pca.explained_variance_ratio_array([0.72962445, 0.22850762])
두개로 95%정도는 설명이 가능한 것 같습니다.
이제 이렇게 pca처리한 iris를 df화 시키는 함수를 만들어보겠습니다.
def get_pd_from_pca(pca_data, cols=['PC1', 'PC2']):
return pd.DataFrame(pca_data, columns=cols)컬럼이 2개일때에만 쓸 수 있는 함수이긴 합니다 ㅎㅎ
iris_pd_pca = get_pd_from_pca(iris_pca)
iris_pd_pca['species'] = iris.target
iris_pd_pca.head()
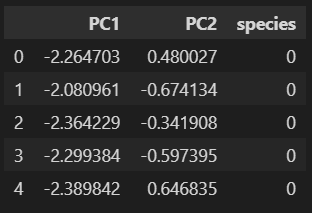
iris.data에서 scaling 시킨 데이터로 pca를 만들었기 때문에 target은 없는 상태였으므로 target데이터를 species 컬럼에 넣어줍니다.
이걸가지고 pairplot을 다시 그려보겠습니다.
sns.pairplot(iris_pd_pca, hue='species', height=5, x_vars=['PC1'], y_vars=['PC2'])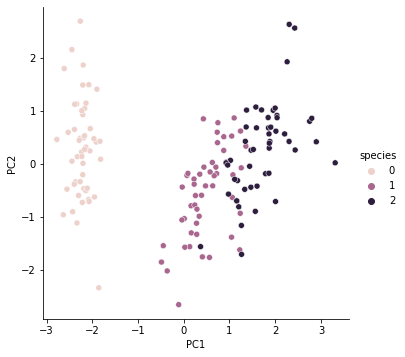
강의에서는 기존 feature축도 같이 넣어서 그려줬네요. 저는 코드로 못짜겠습니다. ㅎㅎ
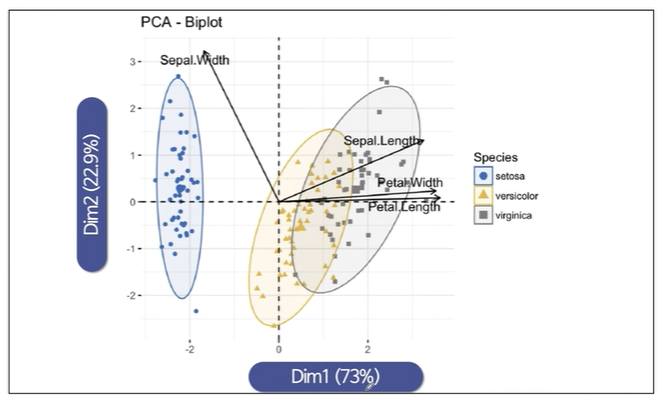
주성분 분석위 위력을 보겠습니다.
4개의 feature를 쓰는게 아니라 2개의 feature를 가지고 73% 23%의 설명력을 가졌다고 하면 꽤 괜찮은 것입니다.
이걸로 machine learning을 돌리면 어떤 결과가 나올까요?
먼저, pca를 쓰기 전 machine learning을 돌려보겠습니다.
RandomForestClassifier를 쓸 거고 cross_validation_score를 통해 5개의 cv로 나누어 진행하고, 여기서 나온 accuracy를 mean값으로 가져와보는 함수를 만들어봅니다.
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
def rf_scores(X, y, cv=5):
rf = RandomForestClassifier(random_state=13, n_estimators=100)
scores_rf = cross_val_score(rf, X, y, scoring='accuracy', cv=cv)
print('Score : ', np.mean(scores_rf))
rf_scores(iris_ss, iris.target) #X는 iris_ss, y는 iris.target이죠.Score : 0.96
X는 iris_ss, y는 iris.target이죠.
이번에는 pca를 가지고 똑같이 적용해보겠습니다.
pca_X = iris_pd_pca[['PC1', 'PC2']]
rf_scores(pca_X, iris.target)Score : 0.9066666666666666
pca후 전체가 95%의 설명률을 가지고 있고 데이터를 100%반영한게 아니라서 score가 떨어지긴 했습니다. 그러나 특성을 줄여서도 머신러닝을 돌릴 수 있다는걸 알게되었습니다.
'머신러닝 > PCA(Principal Component Analysis)' 카테고리의 다른 글
| MNIST using PCA and kNN (1) | 2024.06.04 |
|---|---|
| HAR using PCA (0) | 2024.06.04 |
| PCA - eigenface (0) | 2024.06.01 |
| PCA - wine 데이터 (0) | 2024.06.01 |
| PCA란? (0) | 2024.05.31 |



