Principal Component Analysis (PCA)
- 데이터 집합 내에 존재하는 각 데이터의 차이를 가장 잘 나타내주는 요소를 찾아내는 방법
- 통계 데이터 분석(주성분 찾기), 데이터 압축(차원 감소), 노이즈 제거 등 다양한 분야에서 사용
- 주 성분 분석의 약자로 차원축소(dimensionality reduction)와 변수추출(feature extraction) 기법으로 널리 쓰임
- PCA는 데이터의 분산(variance)을 최대한 보존하면서 서로 직교하는 새 기저(축)을 찾아, 고차원 공간의 표본들을 선형 연관성이 없는 저차원 공간으로 변환하는 기법
- 변수추출(feature extraction)은 기존 변수를 조합해 새로운 변수를 만드는 기법으로 변수선택(feature selection)과 구분할 것
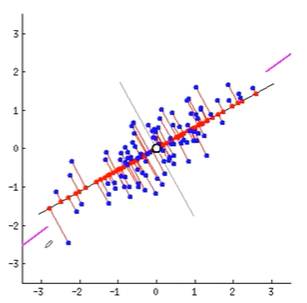
2차원 상에 파란 점으로된 데이터들이 있는데, 1차원으로된 직선에 직교시킨 빨간 점들이 가장 넓게 분포된 순간을 잡아 그 특성으로 가져오겠다는 겁니다. 그러면 분포를 그대로 둔 채, 2차원상의 데이터를 1차원상의 데이터로 가져올 수 있는것이죠. 데이터를 어떤 벡터에 정사영시켜 차원을 낮출 수 있는겁니다.
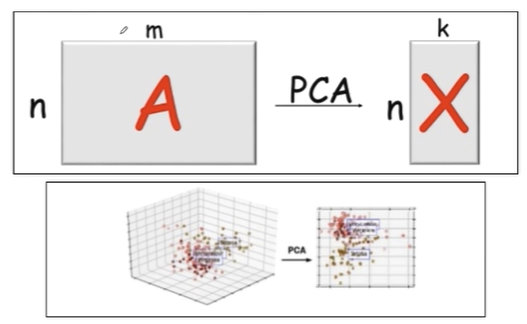
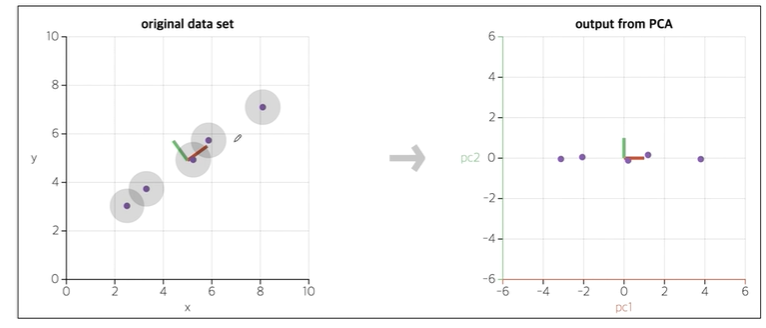
그래서 원본의 데이터가 있다면 PCA를 통해서 아주 차원이 낮은 데이터로 옮기겠다는 뜻입니다.

이제 코드를 통해 실습해보겠습니다.
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
데이터 전처리를 위해 필요한 모듈을 전부 들고옵니다.
numpy에서는 random 모듈을 가져와서 numpy에서 제공하는 dot()함수를 통해 랜덤하게 2by2 행렬과 2by200짜리 행렬을 뽑아내고 그 둘을 곱합니다. 그리고 .T는 transpose시키라는 뜻이죠.
rng = np.random.RandomState(13)
X = np.dot(rng.rand(2, 2), rng.randn(2, 200)).T
#np에서 제공하는 dot()함수를 통해 rng.randn(2,2) 랜덤하게 2 by 2 짜리 행렬을 뽑고, 랜덤하게 2 by 200 짜리 행렬을 뽑습니다.
X.shaperand(2,2)는 2by2로 0부터 1 사이에 있는 값들을 뽑아낸 것입니다.
randn(2, 200)은 2by200으로 표준편차가 1이고 평균이 0인 정규분포(randn)를 따르는 숫자들을 의미합니다.
코드로 각각을 확인해보겠습니다.
rng.rand(2, 2)array([[0.60735693, 0.79695268],
[0.81266616, 0.26598051]])
2 by 2로 0부터1사이에 있는 값들을 뽑아낸겁니다.
rng.randn(2, 200)Output exceeds the size limit. Open the full output data in a text editor
array([[-2.23305285e+00, 1.42026788e-01, 7.42047827e-01,
1.11144113e+00, 4.49767208e-01, -1.43039670e+00,
9.42539772e-01, -5.35146593e-01, -4.58967168e-01,
...
-1.66255235e+00, 1.19085839e+00, -1.81929933e+00,
2.99991138e-01, -3.44396204e-02, -1.67487456e+00,
-1.89409739e-01, -1.00886454e+00, 4.15190466e-01,
-6.18282384e-01, 2.62568229e-01, 3.96449918e-01,
1.14781205e+00, -8.45186532e-01]])
2 by 200으로 표준편차가 1이고 평균이 0인 정규분포(randn)를 따르는 숫자들입니다.
그리고 이 둘을 곱합니다.
np.dot(rng.rand(2, 2), rng.randn(2, 200))Output exceeds the size limit. Open the full output data in a text editor
array([[-1.11411586, 0.26963063, -2.35812802, -1.10004702, 0.46583063,
-2.12906763, -0.73210845, -0.10302759, 0.87991633, -0.48262294,
...
0.52832396, -0.22914893, -1.49277575, -0.92896166, -1.14832974,
-0.29558172, -1.5562435 , 2.05960645, 0.20190503, -0.76372606],
[-0.52915956, 0.23382027, -1.21463222, -0.42126305, 0.24329643,
-1.05972675, -0.33480313, -0.1805496 , 0.37844267, -0.24286828,
...
0.54837685, -0.0397205 , -0.81669638, -0.45671496, -0.38641952,
-0.12675486, -0.8730726 , 1.31156761, 0.33571366, -0.53759776]])
그리고 이 행렬을 transpose시키면
np.dot(rng.rand(2,2), rng.randn(2,200)).TOutput exceeds the size limit. Open the full output data in a text editor
array([[-2.28478921e-02, 9.98220980e-02],
[ 6.26843943e-02, 5.08199149e-01],
...
[ 6.30948828e-02, 2.08009818e-01],
[ 1.79980224e-01, 1.30883314e+00],
[-3.04930351e-01, -1.60677138e+00],
[-1.67220558e-01, -7.99086723e-01],
[ 5.86339218e-02, 6.62195440e-01]])
이렇게 나오게 됩니다.
우리가 흔히들 머신러닝을 돌리던 그 데이터처럼 생겼네요.
scatter 함수를 통해 그려보겠습니다.
plt.scatter(X[:, 0], X[:, 1])
plt.axis('equal')(-2.346839332894307, 2.4400046646752487, -3.859118166687874, 4.08448257499405)
원래 matplotlib로 그래프를 그리면 기본적으로 x축 길이가 더 깁니다. 따라서 이를 보정해줘야 합니다.
이제 pca를 써보도록 하겠습니다.
from sklearn.decomposition import PCA
pca = PCA(n_components=2, random_state=13)
pca.fit(X)PCA(n_components=2, random_state=13)
n_components는 2개의 주 성분으로 나타내달라는 뜻입니다.
이렇게 만들어진 pca에 대해서 확인해보겠습니다.
주 성분이 두개므로 두개의 벡터값이 나와야 합니다.
pca.components_array([[ 0.47802511, 0.87834617],
[-0.87834617, 0.47802511]])
첫행이 하나의 벡터입니다. 첫행의 x축y축을 길이를 통해 벡터를 알 수 있습니다.
행이 두개이므로 X데이터를 설명하는 큰 벡터가 2개입니다. n components를 2로 설정했기 때문입니다.
pca.explained_variance_array([1.82531406, 0.13209947])
설명력이라고 하는데, 첫번째 행이 첫번째 데이터, 두번째 행이 두번째 데이터입니다. 첫번째 행의 벡터가 설명을 잘한다는 뜻입니다. 만약 이걸 비율로 보고싶다면, explained_variance_ratio_를 쓰면 됩니다.
pca.explained_variance_ratio_array([0.93251326, 0.06748674])
이 함수를 쓰면 되고, 첫번째 벡터로 93% 정도로 설명가능하다고 합니다.
시각화하는 방법은 scikit learn에서 튜토리얼에 나오는 아래 함수 코드를 쓰면 됩니다.
def draw_vector(v0, v1, ax=None):
ax = ax or plt.gca()
arrowprops = dict(
arrowstyle = '->',
linewidth = 2,
color = 'black',
shrinkA = 0,
shrinkB = 0
)
ax.annotate('', v1, v0, arrowproprs=arrowprops)이 함수에 대해 간략히 설명하자면, ax=None이 들어오면 ax = plt.gca()가 되는거고, 설정이 들어오면 ax=ax로 그걸 받아서 ax로 쓰라는 뜻입니다.
v1지점부터 v0지점까지 화살표를 그리라는 뜻이고, arrowprops는 원래 dict()형태로 받으라고 한다고 합니다.
원래 annotate함수가 글자를 찍는 함수인데 화살표를 그리는게 목표이기 때문에 ''라고 빈칸으로 두는 것입니다.
plt.scatter(X[:, 0], X[:, 1], alpha=0.4)
for length, vector in zip(pca.explained_variance_, pca.components_):
v = vector * 3 * np.sqrt(length) #3배는 좀 눈에 보이게 키우려고 곱한 것, vector방향으로 설명력(length)만큼의 크기로 v를 그릴것.
draw_vector(pca.mean_, pca.mean_ + v) #pca.mean은 군집의 제일 중앙, 좌표원점이라고 생각하면 됩니다. 거기서부터 v만큼 움직인 곳까지 그림을 그릴 것
plt.axis('equal')
plt.show()vector에 3을 곱한건 눈에 보이게 키우려고 곱한 것이고, vector방향으로 설명력(length)만큼의 크기로 v를 그리라고 했습니다.
pca.mean은 군집의 제일 중앙, 좌표원점이라고 생각하면 됩니다. 거기서부터 v만큼 움직인 곳까지 그림을 그리라고 했습니다.
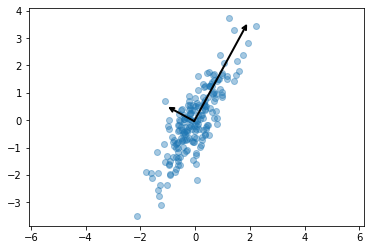
우리의 데이터는 알고봤더니, 저 두 벡터와 그 크기(설명력, length)로 모두 설명할 수 있다고 보는겁니다.
이렇게 데이터의 주성분을 찾은 다음 주축을 변경하는 것도 가능합니다.
제일 중요한 축을 x축으로 봐서 다시 그릴 수 있는겁니다.
x, y일때는 특성 이름이 존재하지만, PC1, PC2의 주성분으로 하면 원래 있던 특성은 사라지고 원 데이터의 새로운 특성을 찾은 것이기 때문에 이름이 없습니다.
그림으로 살펴보면 이런 느낌입니다.

이번에는 방금 전 데이터에서 n_components를 1로 두어보겠습니다.
pca = PCA(n_components=1, random_state=13)
pca.fit(X)
pca.components_array([[0.47802511, 0.87834617]])
네, 이 방향이구요,
pca에서 확인해야할 것들을 다 확인해볼까요?
pca.mean_array([-0.03360413, -0.03527382])
pca.explained_variance_ratio_array([0.93251326])
component 1개만 했을때, 전체 데이터의 93%정도만 반영하네요
이제 원 데이터 X를 transform 시키겠습니다.
X_pca = pca.transform(X)
X_pcaOutput exceeds the size limit. Open the full output data in a text editor
array([[-2.95245605e-01],
[ 5.81990204e-01],
[ 4.94740664e-01],
...
[-4.05753757e-02],
[ 6.77481936e-01],
[-2.13633039e-01]])
fit을 시켰으니 transform 시킵니다. 이제 한 컬럼으로 나오는 것을 확인할 수 있습니다. 즉 주성분이 1개가 된 것입니다.
원래 X데이터는 2열로 되어있었는데 그걸 1열로 줄여버린겁니다. X를 다시 확인해보겠습니다.
XOutput exceeds the size limit. Open the full output data in a text editor
array([[-1.07103225e-01, -3.31411265e-01],
[ 3.61221011e-01, 4.12447007e-01],
...
[ 5.32736506e-01, 4.27820239e-01],
[-4.00361676e-01, -7.88941168e-02]])
하나의 component로 변경했네요 ㅎㅎ 이건 어떻게 된 것인가 scatter로 관찰해보겠습니다.
그 전에 inverse_transform 시켜서 X_new를 만들어야 비교가 가능합니다.
X_new = pca.inverse_transform(X_pca)
X_newOutput exceeds the size limit. Open the full output data in a text editor
array([[-1.74738944e-01, -2.94601664e-01],
...
[-1.35726088e-01, -2.22917579e-01]])
inverse transform은 원래대로 복구하려고 노력한다는 얘기였죠, 즉 2차원상의 1차원의 X였네요.
위의 X데이터랑 살펴보면 원래 데이터와는 다르죠? 어차피 손실되었기 때문입니다 ㅎㅎ
다시 말해서 1차원으로 바꾼 X_pca를 1차원상에 일직선으로 옮겨놓은게 X_new이죠.
말로 해서는 이해가 안갈 수 있으니 그림으로 보여드리겠습니다.
X_new = pca.inverse_transform(X_pca)
plt.scatter(X[:,0], X[:,1], alpha=0.3)
plt.scatter(X_new[:,0], X_new[:,1], alpha=0.9)
plt.grid()
plt.axis('equal')
plt.show()alpha를 줘서 약간 투명도를 줘봤습니다.

저 주황색이 X_new입니다. 이제 이해 가시나요 ㅎㅎ
'머신러닝 > PCA(Principal Component Analysis)' 카테고리의 다른 글
| MNIST using PCA and kNN (1) | 2024.06.04 |
|---|---|
| HAR using PCA (0) | 2024.06.04 |
| PCA - eigenface (0) | 2024.06.01 |
| PCA - wine 데이터 (0) | 2024.06.01 |
| PCA - iris 데이터 (0) | 2024.06.01 |



