1. 교차검증이 필요한 이유
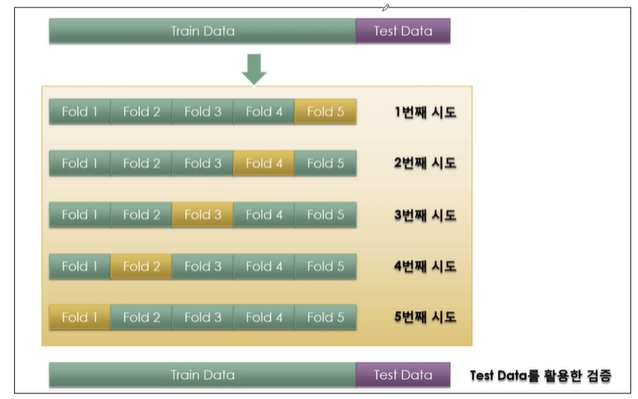
이전에 과적합을 막기위해 train과 test 데이터로 분류한다고 했습니다. 심지어 train 데이터를 또 한 번 분류하는 데 그것이 validation 검증데이터라고 했습니다. 이걸 해보겠습니다.
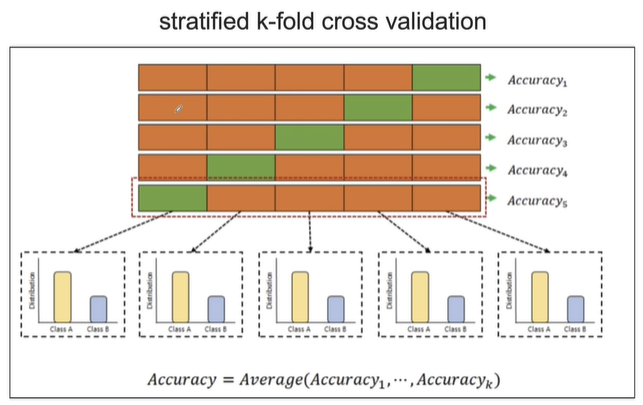
k-fold cross validation이라고 총 train 데이터를 5개로 구분짓고 그 중 4개로 train하고 나머지 하나로 validation 하는 방법을 말합니다.
사진과 같이 5번의 검증하는데 이 때 train 데이터의 accuracy를 모두 평균내서 가져갑니다.
교차 검증을 구현해보겠습니다
2. k-fold cross validation
import numpy as np
from sklearn.model_selection import KFoldX = np.array([
[1,2],[3,4],[1,2],[3,4]
])
y = np.array([1,2,3,4])
X
y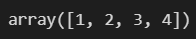
instanciation 시키겠습니다.
kf = KFold(n_splits=2) #몇등분을 할건지 쓰는겁니다. 3,5를 주로 많이 씁니다.
print(kf.get_n_splits(X)) #몇등분 했는지 확인하겠습니다.주의할 점으로 n_splits=를 꼭 주어 몇등분을 할건지 적어야합니다. default값은 5이고 3과 5를 주로 많이 씁니다.

2등분 되었다고 나오네요. kf도 한번 프린트해보겠습니다.
print(kf)
for train_idx, test_idx in kf.split(X):
print(train_idx)
보면 index를 가져온 것이고, 2번3번 인덱스와 0번1번 인덱스로 split한 것이라는 뜻입니다.
이게 의심간다면 한번 더 print해서 test idx도 가져오겠습니다.
for train_idx, test_idx in kf.split(X):
print('Train idx:',train_idx)
print('Test idx:',test_idx)
split을 두번 했으므로, 첫번째 검증에서는 2,3이 train, 0,1이 test인 것이고, 두번째 검증에서는 0,1번이 train, 2,3이 test인 것입니다.
이제 실제 데이터를 보고싶으니까 위의 코드를 약간 수정해보겠습니다.
# 이제 실제 데이터를 보고싶으니까 위의 코드를 약간 수정해보겠습니다.
for train_idx, test_idx in kf.split(X):
print('----idx-----')
print(train_idx,test_idx)
print('----train data----')
print(X[train_idx])
print('----validation data')
print(X[test_idx])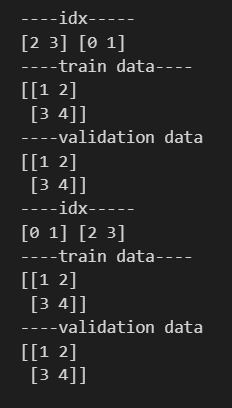
이제 다시 와인 맛 분류로 돌아가보겠습니다. 와인 데이터를 가져오겠습니다.
import pandas as pd
red_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-red.csv'
white_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/winequality-white.csv'
red_wine = pd.read_csv(red_url, sep=';')
white_wine = pd.read_csv(white_url, sep=';')
red_wine['color'] = 1.
white_wine['color'] = 0.
wine = pd.concat([red_wine, white_wine])
와인 맛 분류를 위해 데이터를 정리하겠습니다.
wine['taste'] = [1. if grade>5 else 0. for grade in wine['quality']]
X = wine.drop(['taste', 'quality'], axis=1)
y = wine['taste']
feature와 label로 나눴습니다.
이제 의사결정나무 코드를 가졍겠습니다. pipeline을 안쓴거로 가져와보겠습니다.
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.2, random_state=13
)
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(X_train, y_train)
y_pred_tr = wine_tree.predict(X_train)
y_pred_test = wine_tree.predict(X_test)
print('Train Acc : ', accuracy_score(y_train, y_pred_tr))
print('Test Acc : ', accuracy_score(y_train, y_pred_tr))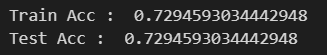
저희가 기존에 썼던 방식으로 얻은 정확도입니다. 그런데 이 정확도가 정말 확실할까요? random으로 나누는 것에 따라 달라진다면 이 정확도를 믿어도 될까요? 여기서 데이터를 저렇게 분리하는 것이 최선일까요?
따라서 저 accuracy를 어떻게 신뢰할 수 있는지를 확인하기 위해서 생겨난 것이 k-folding을 사용한 cross validation이 생겨난 겁니다. 보통 5-fold를 주로 사용합니다.
from sklearn.model_selection import KFold
kfold = KFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cross validation 했다고 cv를 뒤에 붙였습니다.
현재 tree와 kfold가 각각 설정되었지 서로 연결되지는 않았기 때문에, 이 둘을 연결시켜주겠습니다.
우리는 X데이터를 가져있고 kfold를 통해서 X_train_idx와 test_idx를 가져올 수 있습니다.
length를 우선 확인해보겠습니다.
for train_idx, test_idx in kfold.split(X):
print(len(train_idx), len(test_idx))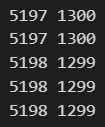
한개 두개 정도는 정수로 안나눠지기 때문에 5197개로 훈련하고 1300개로 검증하는 5가지로 나뉘었습니다.
그러면 학습은 어떻게 시킬까요? kfold에서 idx들을 가져와서 X[idx]들로 데이터를 나눌겁니다.
for train_idx, test_idx in kfold.split(X):
X_train = X.iloc[train_idx] #X_train은 kfold가 split해준 X의 idx를 X에[]로 할당하면 X_train이 되는겁니다.
X_test = X.iloc[test_idx]
#학습하려면 label 데이터도 필요합니다.
y_train = y.iloc[train_idx]
y_test = y.iloc[test_idx]
#이렇게 5번의 for문이 돌면서 train test 데이터가 만들어집니다.
#이제 5번의 검증과 정확도 확인을 위해 tree에 fit시켜주겠습니다.
wine_tree_cv.fit(X_train, y_train)
pred = wine_tree_cv.predict(X_test)
print(pred)
X_train은 kfold가 split해준 X의 idx를 X에[]로 할당하면 X_train이 되는겁니다. 위 처럼 5번의 for문이 돌면서 train test 데이터가 만들어집니다. 그리고 5번의 검증과 정확도 확인을 위해 tree에 fit시켜주었습니다.
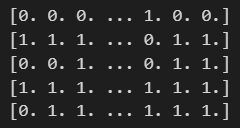
5번의 무언가를 한 것으로 보입니다. validation data에 대해서 accuracy가 얼마나 나오는지 살펴보겠습니다.
for train_idx, test_idx in kfold.split(X):
X_train = X.iloc[train_idx]
X_test = X.iloc[test_idx]
y_train = y.iloc[train_idx]
y_test = y.iloc[test_idx]
wine_tree_cv.fit(X_train, y_train)
pred = wine_tree_cv.predict(X_test)
print(accuracy_score(y_test, pred))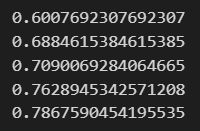
내 모델이 하나의 accuracy가 아닐 수 있겠다는 걸 알 수 있습니다. 이 기록을 보관하고 싶으므로 빈 리스트에 이걸 담을겁니다.
cv_accuracy = []
for train_idx, test_idx in kfold.split(X):
X_train = X.iloc[train_idx]
X_test = X.iloc[test_idx]
y_train = y.iloc[train_idx]
y_test = y.iloc[test_idx]
wine_tree_cv.fit(X_train, y_train)
pred = wine_tree_cv.predict(X_test)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy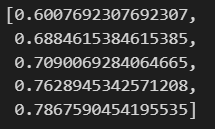
각 acc의 분산이 크지 않다면, 평균값을 보겠습니다.
np.mean(cv_accuracy)
3. Stratified k-fold cross validation
이번에는 stratified k-fold를 쓰는 방법에 대해 알아보겠습니다. stratify 기능을 기억하십니까?
stratify는 지정한 Data의 비율을 유지합니다. 예를 들어, Label Set인 y가 25%의 0과 75%의 1로 이루어진 Binary Set일 때, stratify=y로 설정하면 나누어진 데이터셋들도 0과 1을 각각 25%, 75%로 유지한 채 분할됩니다.
from sklearn.model_selection import StratifiedKFold
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
cv_accuracy = []
for train_idx, test_idx in skfold.split(X, y): #skfold로 변경하면 X뿐만 아니라 어떤걸 기준으로 stratify를 적용할건지 적어야 합니다. 따라서 y도 같이 변수로 넣어줍니다.
X_train = X.iloc[train_idx]
X_test = X.iloc[test_idx]
y_train = y.iloc[train_idx]
y_test = y.iloc[test_idx]
wine_tree_cv.fit(X_train, y_train)
pred = wine_tree_cv.predict(X_test)
cv_accuracy.append(accuracy_score(y_test, pred))
cv_accuracy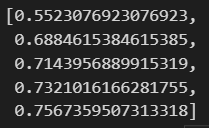
np.mean(cv_accuracy)
평균이 더 내려갔습니다. 즉 모델의 성능이 예상보다 좋지않구나 라는걸 알 수 있습니다.
이렇게 for문으로 처리하면 코드가 길어지기 때문이 이 또한 모듈이 있습니다. cross_val_score라는 모듈입니다.
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=2, random_state=13)
#cross validation을 보다 간단하게 진행해보겠습니다.
cross_val_score(wine_tree_cv, X, y, scoring=None, cv=skfold)
tree 넣어주고 데이터 넣어주고 cv는 skfold를 써라라고 명령하면 array를 뱉습니다.
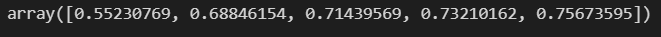
np.mean(cross_val_score(wine_tree_cv, X, y, scoring=None, cv=skfold))
그러나 가끔은 원시적인 접근이 필요할 때가 있어서 for문도 돌려볼 줄 알아야합니다.
대체로 cross_val_score()를 주로 쓰긴 합니다...
max_depth를 5로 바꿔서 봐보겠습니다.
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=5, random_state=13)
cross_val_score(wine_tree_cv, X, y, scoring=None, cv=skfold)
평균이 더 내려갔습니다. 즉 모델의 성능이 예상보다 좋지않구나 라는걸 알 수 있습니다.
이렇게 for문으로 처리하면 코드가 길어지기 때문이 이 또한 모듈이 있습니다. cross_val_score라는 모듈입니다.
위 코드를 함수로 만들어두겠습니다.
def skfold_dt(depth): #skfold를 하는 decision tree 알고리즘
from sklearn.model_selection import cross_val_score
skfold = StratifiedKFold(n_splits=5)
wine_tree_cv = DecisionTreeClassifier(max_depth=depth, random_state=13)
print(cross_val_score(wine_tree_cv, X, y, scoring=None, cv=skfold))
skfold_dt(3)
train score와 함께 보고싶다면 cross_validate라는 모듈로 같이 가져옵니다.
from sklearn.model_selection import cross_validate
cross_validate(wine_tree_cv, X, y, scoring=None, cv=skfold, return_train_score=True)
여기서 return_train_score를 True로 주면 됩니다.
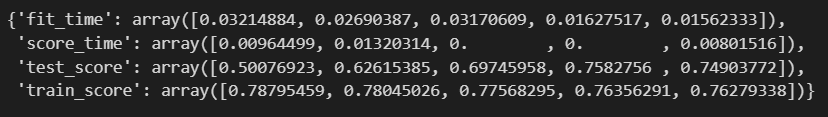
train 에서보다 test가 28%의 차이가 나는 과적합 현상이 보입니다.
'머신러닝 > Preprocessing' 카테고리의 다른 글
| GridSearchCV을 통한 하이퍼파라미터 튜닝 (1) | 2024.03.07 |
|---|---|
| Scikit-learn Pipeline으로 만들기 (1) | 2024.03.05 |
| Decision Tree를 이용한 Wine 데이터 분석_이진분류 (0) | 2024.03.04 |
| Decision Tree를 이용한 Wine 데이터 분석_데이터 처리 (1) | 2024.03.01 |
| Decision Tree를 이용한 Wine 데이터 분석_데이터 확인 (1) | 2024.02.29 |



