PIMA 인디언은 멕시코와 미국에 걸쳐살고있던 인디언 부족이라고 합니다. 1950년대까지 PIMA 인디언은 당뇨가 없었습니다. 그런데 20세기 말, 단 50년만에 인구의 50%가 당뇨에 걸렸고 50년만에 50%의 인구가 당뇨에 걸렸습니다.
원래 데이터는 Kaggle에 있습니다. 이 데이터를 pinkwink github에서 가져오겠습니다.
각 컬럼에 대해 이야기하자면 다음과 같습니다.

import pandas as pd
PIMA_url = 'https://raw.githubusercontent.com/PinkWink/ML_tutorial/master/dataset/diabetes.csv'
pima = pd.read_csv(PIMA_url)
pima.head()
pima 데이터를 좀 살펴보겠습니다.
pima.info()
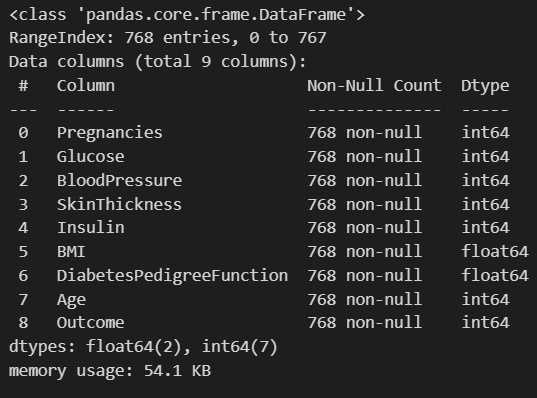
총 768개의 데이터가 있고 전부 수치형 데이터네요.
안전하게 데이터를 처리하기 위해 전부 float형태로 바꿔보겠습니다.
pima = pima.astype('float')
pima.info()
머신러닝 전에 각 컬럼별 상관관계를 확인해보겠습니다.
pima.corr()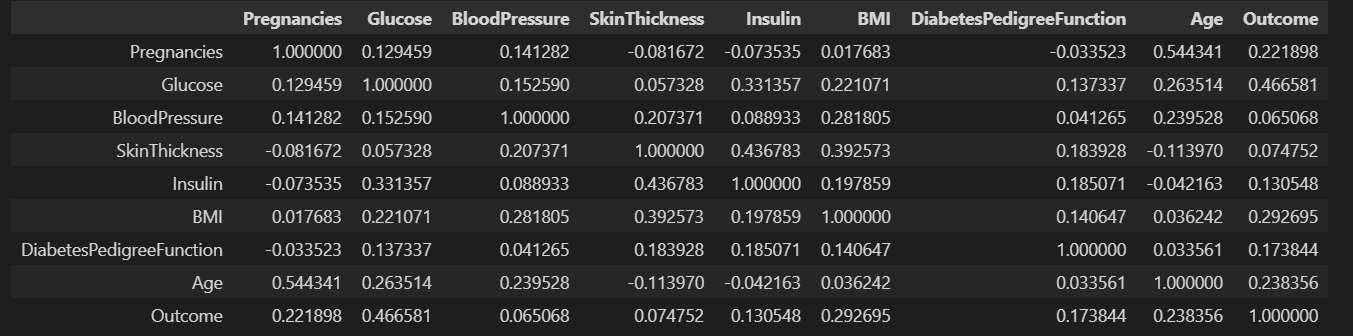
저번시간처럼 이걸 heatmap으로 보기쉽게 그려볼게요
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline #주피터 노트북에서 주로 쓰는 코드
plt.figure(figsize=(12, 10))
sns.heatmap(pima.corr(), annot=True, cmap='YlGnBu')
plt.show()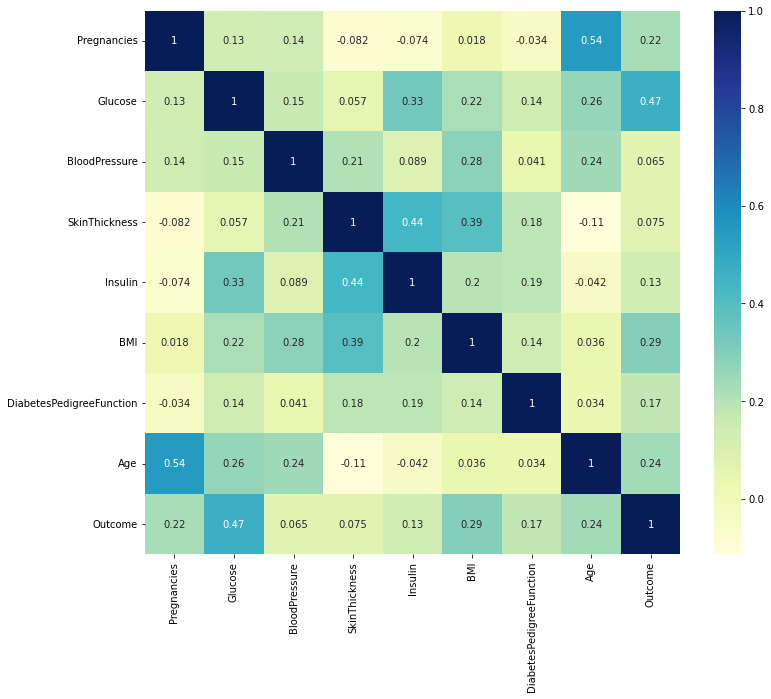
outcome과 관련있는 것들을 찾아보면 pregnancies, glucose, BMI, Age 정도가 있네요... 높은 수치는 아니지만...
그런데 0이 있습니다.
pima==0
(pima==0).astype(int)이렇게하면 false는 0, true는 1이 됩니다.
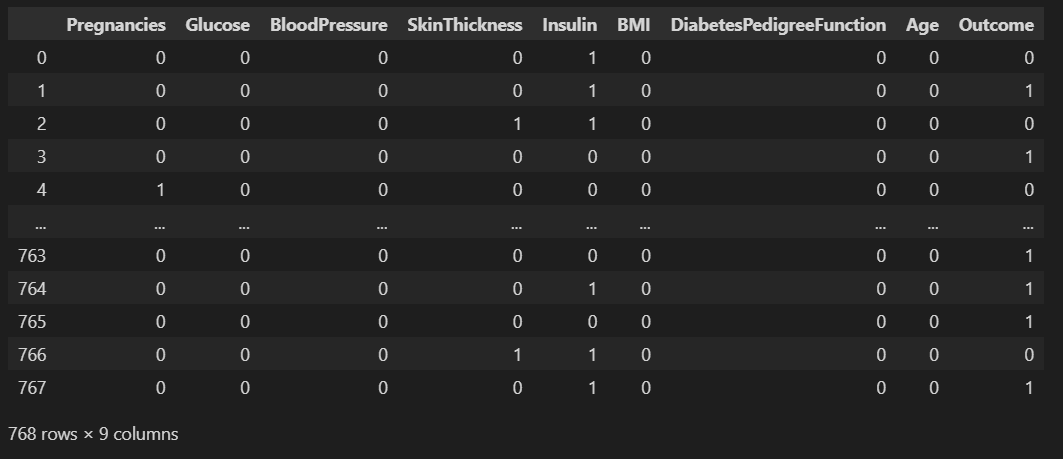
이거를 sum시켜보겠습니다. 그러면 true의 갯수가 나오겠죠.
(pima==0).astype(int).sum()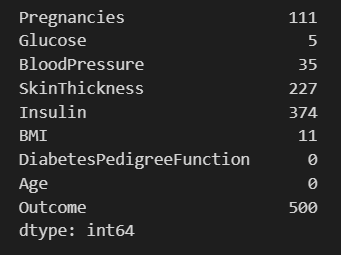
이 0은 null은 아니라서 위 코드가 생각보다 자주 쓰일겁니다.
그런데 혈압이 0인 데이터, 체지방 지수가 0인 데이터가 있다고합니다.
절대로 0이 될 수 없는 데이터를 가져와보겠습니다.
null값이라면 앞뒤 데이터의 중간값이나 부드럽게 넘어갈 수 있는 데이터로 가져오겠지만 0은 수치라서 상당히 힙듭니다.
특히 의학적 지식과 PIMA 인디언에 대한 정보가 없으므로 일단 평균값으로 대체하겠습니다.
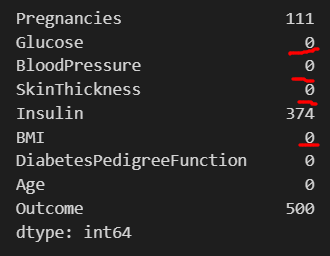
zero_feature = ['Glucose', 'BloodPressure', 'SkinThickness', 'BMI']
pima[zero_feature] = pima[zero_feature].replace(0, pima[zero_feature].mean()) #이렇게하면 해당 열의 평균값으로 대체합니다.
(pima==0).astype(int).sum()파이썬의 좋은점이죠. 열 전체를 replace 시킬 수 있고 replace 대상에 한줄의 코드만으로 각 열마다의 평균값을 넣을 수 있습니다.
이제 데이터를 나누겠습니다.
from sklearn.model_selection import train_test_split
X = pima.drop(['Outcome'], axis=1)
y = pima['Outcome']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2,
#당뇨냐 아니냐에 대한 구조를 그대로 가져가라고 하겠습니다.
stratify=y,
random_state=13)
여기서 저번시간에 썼던 stratify를 적용해보겠습니다. train과 test에 당뇨냐 아니냐에 대한 비율을 그대로 가져가라고 하는 것입니다.
*strafity란?
stratify 매개변수는 데이터를 분할할 때, 분할된 데이터셋이 원본 데이터셋의 클래스 비율을 유지하도록 보장하는 역할을 합니다.이 매개변수를 사용하면 훈련 및 테스트 데이터셋이 원본 데이터셋과 동일한 클래스 비율을 갖게 됩니다. 이는 특히 클래스가 불균형하게 분포되어 있을 때 중요합니다. 예를 들어, 100개의 샘플이 있는데 90개가 클래스 A에, 10개가 클래스 B에 속한다고 가정해 봅시다. 이런 경우, stratify=y를 사용하여 데이터를 분할하면 훈련 및 테스트 세트 각각에 클래스 A와 클래스 B의 비율이 유지됩니다.
pipeline을 만들어주겠습니다. 저번시간처럼 Standard Scaler를 쓰겠습니다.
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression
estimate = [
('scaler', StandardScaler()),
('clf', LogisticRegression(solver='liblinear', random_state=13))
]
pipe_lr = Pipeline(estimate)
pipe_lr.fit(X_train, y_train)fit시켜주고, predict 시키겠습니다. 이번에는 test 데이터만 해보겠습니다.
pred = pipe_lr.predict(X_test)
이제 model evaluation때 배웠던 수치들을 전부 확인해보겠습니다.
from sklearn.metrics import accuracy_score, recall_score, precision_score, roc_auc_score, f1_score
print('Accuracy : ', accuracy_score(y_test, pred))
print('Recall : ', recall_score(y_test, pred))
print('Precision : ', precision_score(y_test, pred))
print('AUC score : ', roc_auc_score(y_test, pred))
print('f1 score : ', f1_score(y_test, pred))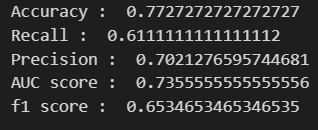
그러나 이 수치들은 수치 자체를 평가할 순 없습니다.
총 컬럼이 8개였으므로 x1 x2 x3, ..., x8 이고 theta1, theta2, ..., theta8으로 총 8개의 변수가 사용된겁니다.
어떤 변수가 가중치가 큰지 확인해보기 위해서 pipe_lr의 clf의 coef_를 확인해보겠습니다.
pipe_lr['clf'].coef_
list가 두개네요... [0]을 붙여서 때고 column이름을 붙여줘야겠습니다.
coef = list(pipe_lr['clf'].coef_[0])
label = list(X_train.columns)
coef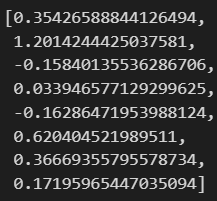
이제 중요한 feature에 대해 map으로 그려보겠습니다.
features = pd.DataFrame(columns=label, data=coef) 이건 안되네요..list는 열에만 넣어집니다.
그래프에 중요도 순서대로 나타나기 위해 importances 순으로 나열해보겠습니다.
features = pd.DataFrame({'Features':label, 'importance':coef})
features.sort_values(by=['importance'], ascending=True, inplace=True)
features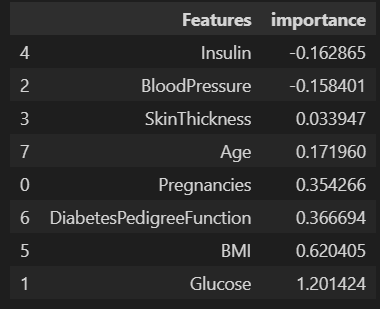
그래프에 color을 입히기 위해 column을 만들어보겠습니다.
features['positive'] = features['importance']>0
features
그래프에 쉽게 표기하기 위해 set_index 시켜줍니다.
features.set_index('Features', inplace=True)
features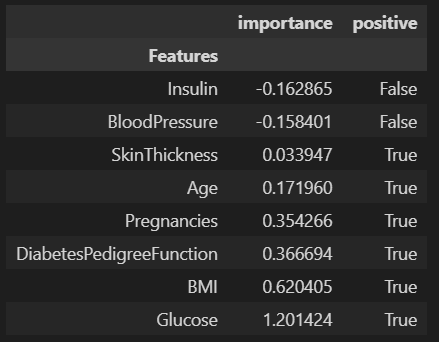
이제 map을 그려보겠습니다.
features['importance'].plot(kind='barh',
figsize=(11,6),
color=features['positive'].map({True:'blue', False:'red'}) #barh에 color를 부여하기 위한 옵션입니다.
)
plt.xlabel('importance')
plt.show()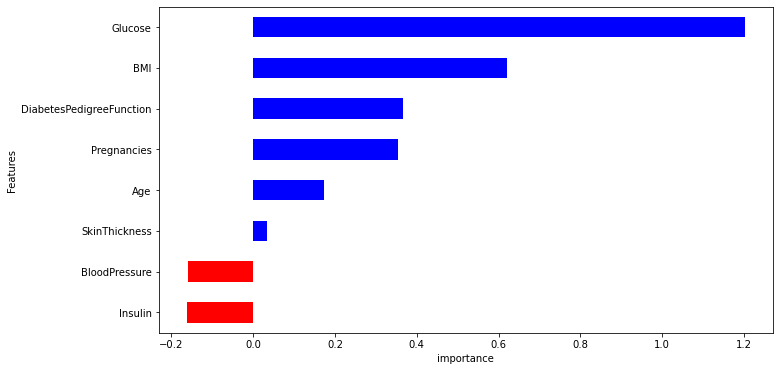
네, 포도당, BMI등은 당뇨에 영향을 미치는 정도가 높습니다.
혈압은 예측에 부정적인 영향을 줍니다.
연령이 BMI보다 출력 변수와 더 관련되어 있었지만, 모델은 BMI와 Glucose에 더 의존합니다.
'머신러닝 > Logistic Regression' 카테고리의 다른 글
| Precision(정밀도)과 Recall(재현율)의 Trade off (0) | 2024.05.07 |
|---|---|
| Logistic Regression - PIMA 인디언 당뇨병 예측 (0) | 2024.05.07 |
| Logistic Regression - 실습 (0) | 2024.05.06 |
| Logistic Regression - 이론 (0) | 2024.05.06 |



